GEPIA
- 科技
- 1周前
- 65
快速启动:
在“输入基因名称”字段中输入一个基因符号或基因ID( ID),然后单击“GoPIA!”按钮搜索感兴趣的基因。

微分表达式分析
该特性允许用户在给定的数据集上应用自定义统计方法和阈值来动态获取差异表达的基因及其染色体分布。
参数
数据集:选择感兴趣的癌症类型。
染色体分布
选择“高表达”、“低表达”或“两者”作为染色体分布图。
微分法:选择一种差异分析方法。
\x日志2FC_截断
设置自定义折叠-更改阈值。
Q值截止
设置自定义Q值阈值。
百分比截止
设置自定义百分比阈值。
对于方差分析(ANOVA)和LIMMA(LIMMA)选项,基因具有较高的比2FC值和低于预设阈值的Q值被认为是差异表达的基因.
在前10名中,对数较高的基因2FC值和高于阈值的百分比值被认为是过度表达的基因;因此,只有过表达的基因才会出现在列表和染色体图中。
结果
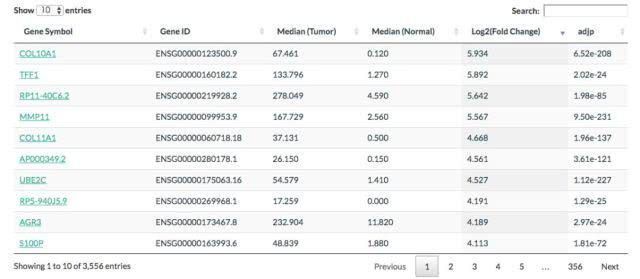
差异基因
单击“列表”按钮:GEPIA将生成一个差异表达基因列表[默认情况下,该列表按日志的降序排列。2基于输入参数。
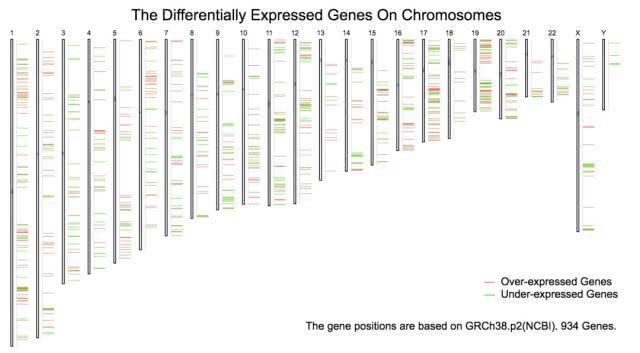
染色体分布图
点击“绘图”按钮:GEPIA将生成染色体分布图。染色体上高表达的基因用红线标记,而低表达的基因被标记为绿色。
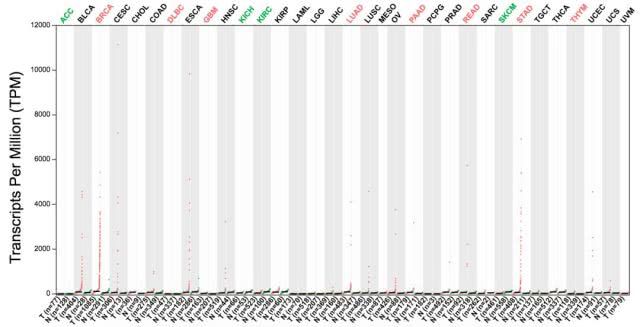
DIY表达
GEPIA根据选定的数据集和按癌症类型或病理分期的统计方法绘制特定基因的表达谱。详情如下:
基因表达谱
GEPIA在多个癌症类型和配对正常样本中产生点图分析基因表达,每个点代表一个不同的肿瘤或正常样本。
参数
基因
输入感兴趣的基因。
数据集/组织顺序:在“”字段中选择感兴趣的癌症类型,然后单击“Add”或“All”,在“组织顺序”字段中构建数据集列表。手动输入由逗号划分的癌症类型(例如ACC、BRCA、BLCA)也是可以接受的。图的x轴将遵循数据集的顺序。
测井标尺:选择使用线性还是日志
TPM+1)转换表达式数据进行绘图。
匹配正常数据:在绘图中,选择“TCGA法线+GTEx法线”、“只有TCGA法线”或“None”作为匹配的正常数据。[用于微分分析的匹配正常样本为“TCGA正常+GTEx正态”]
地块宽度
设置自定义绘图宽度。
微分法
统计方法用于差异基因表达分析。
\x日志2FC_截断
设置自定义折叠-更改阈值。
Q值截止
设置自定义Q值阈值。
百分比截止
设置自定义百分比阈值。
对于方差分析(ANOVA)和LIMMA(LIMMA)选项,基因具有较高的比2FC值和低于预设阈值的Q值被认为是差异表达的基因.
在前10名中,对数较高的基因2FC值和高于阈值的百分比值被认为是过度表达的基因.
结果
点击“绘图”按钮:GEPIA将根据用户的自定义输入参数生成一个基因表达概要文件。
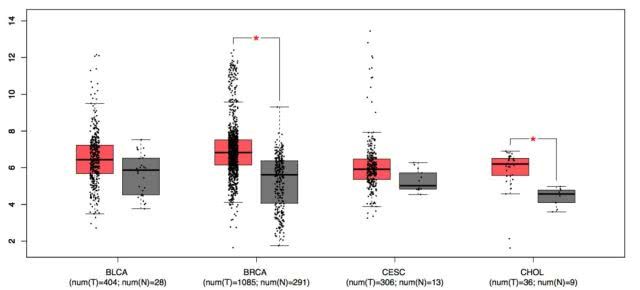
盒样地
GEPIA生成带有抖动的方框图,用于比较几种癌症类型中的表达(为了获得最佳的视觉质量,我们推荐1-4种癌症类型)。
参数
基因
输入感兴趣的基因。
数据集选择/数据集
:在“数据集选择”字段中选择感兴趣的癌症类型,然后单击“添加”在“”字段中生成数据集列表。手动输入由逗号划分的癌症类型(例如ACC、BRCA、BLCA)也是可以接受的。图的x轴将遵循数据集的顺序。
肿瘤颜色:设置肿瘤数据集的框色。
正常颜色:设置普通数据集的框色。
测井标尺:选择使用线性还是日志
TPM+1)转换表达式数据进行绘图。
抖动尺寸:设置整个框内抖动的大小。
差分阈值:
\x日志2FC_截断
设置自定义折叠-更改阈值。
P值截止:设置自定义p值阈值。
匹配正常数据:选择“TCGA法线+GTEx法线”、“只有TCGA法线”进行差异分析和绘图。
这里的差异分析基于选定的数据集。(“TCGA肿瘤与TCGA正常+GTEx正常”或“TCGA肿瘤与TCGA正常”)。差异分析的方法是单向方差分析,以疾病状态(肿瘤或正常)作为变量计算差异表达:
表达式数据是第一个日志。2(TPM+1)变换用于微分分析和日志2FC被定义为中位(肿瘤)-中位数(正常)。
高log基因2FC值和低于预设阈值的Q值被认为是差异表达的基因.
结果
点击“绘图”按钮:GEPIA将显示一个基于用户自定义输入参数的基因表达框图。
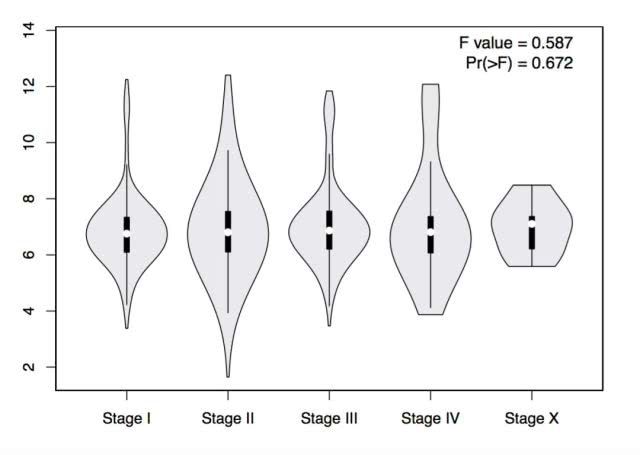
基于病理分期的剖面图
这一特征根据病人的病理阶段生成表达小提琴的情节。
参数
基因
输入感兴趣的基因。
数据集选择/数据集:在“数据集选择”字段中选择一个或多个感兴趣的癌症类型,然后单击“添加”以在“数据集”字段中生成数据集列表。手动输入由逗号分割的癌症类型(例如Coad、Read)也是可以接受的。
测井标尺:选择使用线性还是日志
TPM+1)转换表达式数据进行绘图。
使用主要阶段:选择病理主要阶段或亚阶段进行绘图。
情节色彩
在情节中设置小提琴的颜色。
差异基因表达分析方法为单向方差分析,以病理分期为变量计算差异表达:
表达式数据是第一个日志。2(TPM+1)转换为微分分析。
注意:如果在某一阶段只存在一个样本,该样本将被GEPIA过滤掉,以提高情节的视觉质量。
结果
点击“绘图”按钮:GEPIA将生成一个基于用户自定义输入的基因表达阶段图。
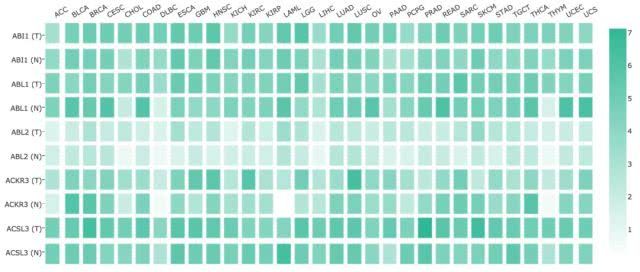
多基因表达比较
此特性提供基于给定基因列表的表达矩阵图。每个块中的颜色密度表示一个基因在给定组织中的中位表达值,通过所有块的最大中值表达值进行归一化。同一肿瘤或正常组织中的不同基因可以在一个地方进行比较。
参数
基因表
输入感兴趣的基因列表。人工输入逗号分裂的基因。
ERBB 2,EGFR)也是可以接受的。
数据集/组织顺序:在“”字段中选择感兴趣的癌症类型,然后单击“Add”或“All”,在“组织顺序”字段中构建数据集列表。此外,手动输入由逗号划分的癌症类型(例如ACC、BRCA、BLCA)也是可以接受的。
测井标尺:选择使用线性还是日志
TPM+1)转换表达式数据进行绘图。
匹配正常数据:选择“只有TCGA肿瘤”、“TCGA肿瘤+TCGA正常+GTEx正常”或“TCGA肿瘤+TCGA正常”进行绘图。
结果
单击“绘图”按钮:GEPIA将根据用户自定义输入参数生成表达式矩阵图。
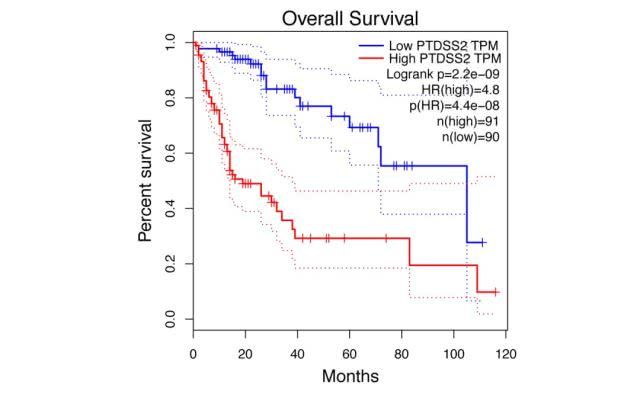
生存分析
GEPIA根据基因表达进行整体生存(OS)或无疾病生存(DFS,也称为无复发生存和RFS)分析。GEPIA采用对数秩检验,即-Cox检验,用于假设检验.可以调整队列阈值,也可以使用基因对。COX比例风险比和95%置信区间信息也可以包含在生存图中。最与病人生存相关的基因可以被搜索。
参数
生存地块
基因
输入感兴趣的基因。
基因归一化
在“基因”领域设置用于正常化的基因。
方法:选择OS或DFS生存方法。
轴心单位:选择月份或日单位进行绘图。
数据集选择/数据集:在“数据集选择”字段中选择一个或多个感兴趣的癌症类型,然后单击“添加”以在“数据集”字段中生成数据集列表。此外,手动输入的癌症类型划分为逗号(例如Coad,读)也是可以接受的。
颜色反转选择是否反转默认颜色。
群截止:选择一个合适的表达阈值来分割高表达和低表达的队列。
截止-高(%):表达水平高于此阈值的样本被认为是高表达群体.
截止-低(%)
表达水平低于此阈值的样本被认为是低表达群体.
最重要的生存基因
数据集选择:选择感兴趣的癌症类型。
方法:选择OS或DFS生存方法。
群截止:选择合适的表达阈值来分割高表达和低表达的队列。
结果
生存地块
单击“绘图”按钮:GEPIA将根据用户自定义输入参数生成生存图。
最重要的生存基因
点击“列表”按钮:GEPIA将生成一个列表,列出在癌症类型下最重要的100种存活基因。
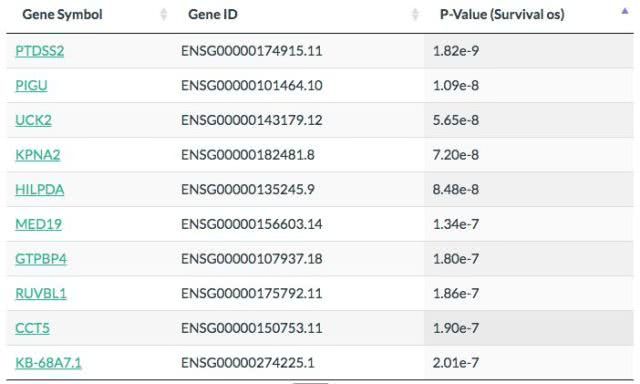
相似基因检测
这一功能识别了与输入基因和所选数据集具有相似表达模式的基因列表。
参数
基因
输入感兴趣的基因。
顶#相似基因:列出最相似的基因。(最高限额:200)
TCGA肿瘤/TCGA正常/GTEx/表达数据集:在“TCGA肿瘤”、“TCGA法线”或“GTEx”字段中选择感兴趣的癌症类型,然后单击“Add”在“二手表达式数据集”字段中构建数据集列表。此外,手动输入由逗号分割的癌症类型(例如Coad肿瘤、读肿瘤)也是可以接受的。相似的基因是根据数据集列表来检测的。
结果
点击“列表”按钮:GEPIA将生成一个由相关系数(PCC)排序的具有相似表达模式的基因列表。
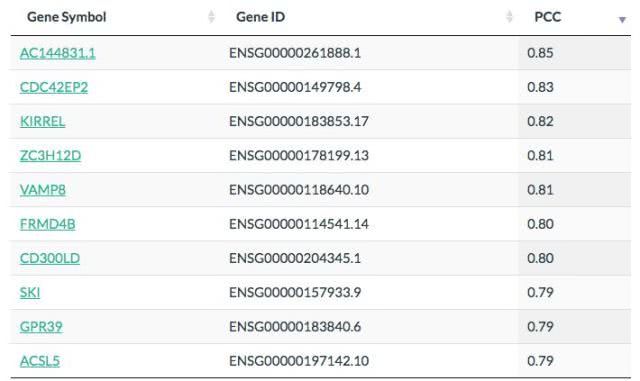
相关性
该函数利用、和等方法对给定的TCGA和/或GTEx表达数据进行成对基因表达相关分析。一个基因可以被另一个基因规范化。
GEPIA使用非对数标度进行计算,并使用对数标度轴进行可视化。
参数
基因A
输入感兴趣的A基因。[x轴]
B基因
输入感兴趣的B基因。[y轴]
基因归一化
设置用于A和B基因正常化的基因。
相关系数
计算相关系数的方法。
TCGA肿瘤/TCGA正常/GTEx/表达数据集
:在“TCGA肿瘤”、“TCGA法线”或“GTEx”字段中选择感兴趣的癌症类型,然后单击“Add”在“二手表达式数据集”字段中构建数据集列表。此外,手动输入由逗号分割的癌症类型(例如Coad肿瘤、读肿瘤)也是可以接受的。相关分析基于数据集列表。
结果
点击“绘图”按钮:GEPIA将生成一个相关分析结果的散点图。
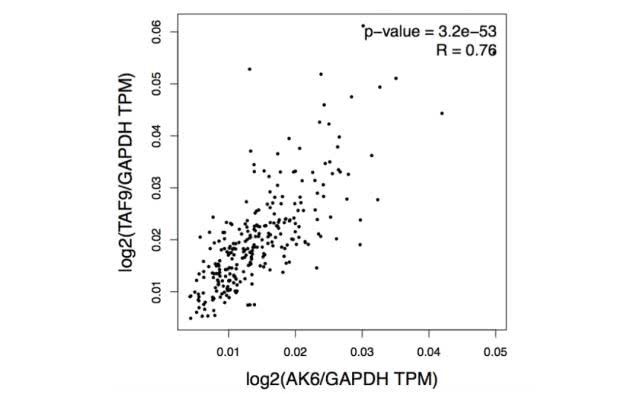
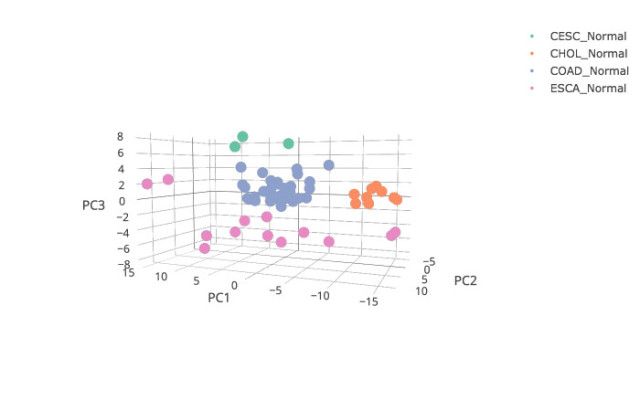
降维
GEPIA使用自定义的TCGA和/或GTEx表达数据对给定的基因列表进行主成分分析(PCA)。首先,GEPIA给出了前三个主成分(PC)的三维图,并为每台PC解释的差异生成一个条形图。第二,GEPIA提供了基于用户指定PC的2D或3D绘图。
参数
第一步
基因表
输入感兴趣的基因列表。人工输入逗号分裂的基因。
ERBB 2,EGFR
也是可以接受的。
测井标尺:选择使用线性还是日志
TPM+1)转换表达式数据进行绘图。
比例尺
选择是否使用Z分数代替表达水平作为PCA分析的输入。
TCGA肿瘤/TCGA正常/GTEx/表达数据集:在“TCGA肿瘤”、“TCGA法线”或“GTEx”字段中选择感兴趣的癌症类型,然后单击“Add”在“二手表达式数据集”字段中构建数据集列表。此外,手动输入由逗号分割的癌症类型(例如Coad肿瘤、读肿瘤)也是可以接受的。主成分分析基于数据集列表。
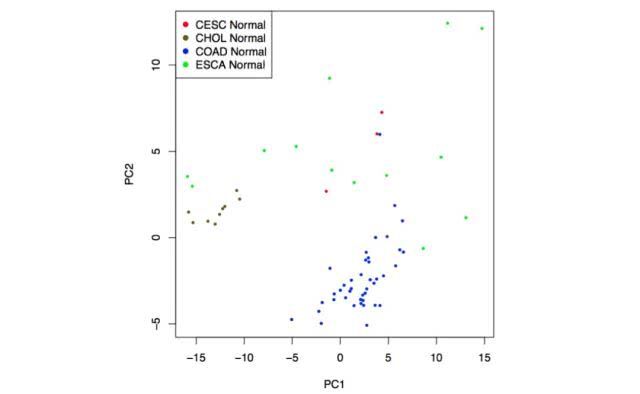
第二步
PC选择:为2D(设置x和y值)或3D散点图(设置x、y和z值)选择PC。
结果
第一步
单击“列表”按钮:GEPIA将根据相关分析结果生成三维散点图和方差分布。
通过单击右上角的照相机图标,3D图形可以保存为.png文件。
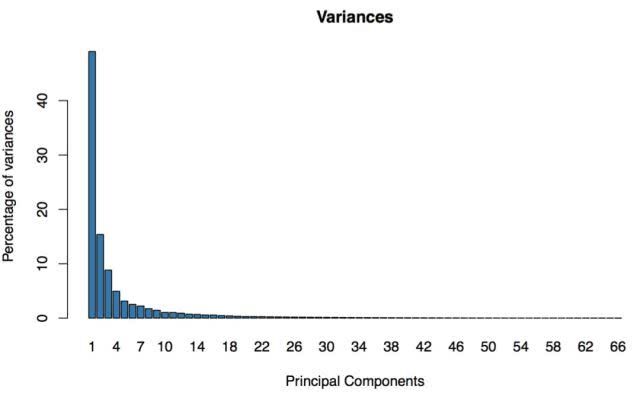
第二步
点击“绘图2D”按钮:GEPIA将生成一个2D散点图的相关分析结果。
点击“绘图3D”按钮:GEPIA将生成3D散点图的相关分析结果。
结果下载:
通过单击结果附近的按钮,可以获得PDF和SVG下载。
编辑结果:
下载的PDF和SVG情节可以由编辑。下面是一个教程视频:








