陪你学·生信十一、构建系统发育树
- 教育
- 40秒前
- 88
- in Makes Sense-
in the Light of
—— (1973)
这个推送主要是系统发育树相关内容(内容很多,但这篇文章并不包含多基因级联建树,挖个坑下次写。我将一些tips和操作示例写在了可以划动的模块里,让整篇推送看起来结构清晰,如果不想看可以跳过划动部分)。上一个推送的多序列比对([陪你学·生信]十、编辑对多序列比对结果[陪你学·生信]九、多序列比对- (MSA))是系统发育分析的基础。
文章有点长,先放个流程图便于理清思路。
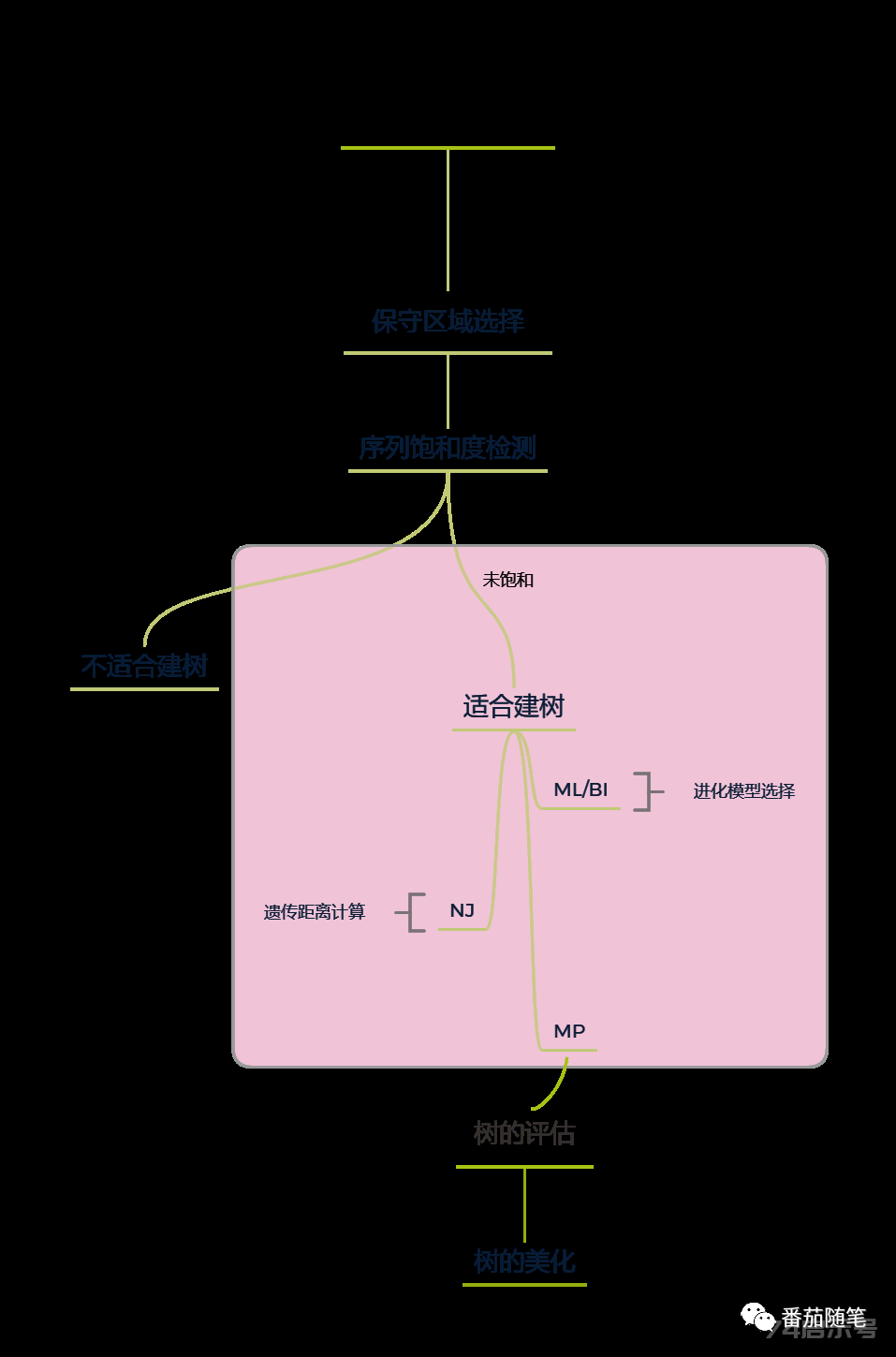
一、系统发育树能分析什么
系统发育分析是根据生物核苷酸/蛋白序列的相似程度(进行MSA)将其归结成分支和簇,从而推测一组基因或蛋白质或生物间的进化关系。当然,系统发育分析的前提是进化论。
实验中系统发育分析常见目的有:物种鉴定,基因功能鉴定,追溯基因起源等。
二、准备序列数据时需要考虑的问题
1. 建什么树?用什么序列?
(1)核苷酸序列nt还是氨基酸序列aa
系统发育分析所选的序列应包含足够的信息,全基因组序列包含信息多,但是全基因组序列的分析麻烦、耗时长;且有的时候研究对象就是某一个基因或者蛋白。所以多数情况下,我们使用基因或蛋白序列建树。
可以的话,选择的这个基因在基因组的拷贝数最好为1,这样避免了旁系同源基因建树的影响。有的生物基因组小,比如病毒,建立物种树有时可以用全基因组序列(nt)进行系统发育分析(如果是蛋白序列同源性分析当然是aa序列,如果是非编码区序列分析当然是nt序列)。
对于可以用aa或者nt序列分析的实验材料,没有标准的答案。如果DNA序列之间的一致度高于70%或进化距离很近用nt序列更合适,因为nt序列的比对已经很整齐了,还可以保留同义突变;如果亲缘关系较远,用aa更合适,MSA时可以使保守区域对齐。也可以两个都试一试,可以加入已知物种/进化关系的同源分子做对照,看哪个结果更符合已知事实。
之前看过一篇论文,说目前发表的文章中构建的系统发育树多数都重复不出来。建树很“主观”,受到序列的选择、MSA、比对后剪辑、建树算法和参数等影响。所以在实验得到一个相对准确和满意的发育树后,最好记录下操作过程和参数。
(2)做的是基因树 (gene tree) 还是物种树 ( tree)
同源基因是指由共同祖先基因衍生的基因,主要有三种关系:直系同源、旁系同源、异源同源。
如果在建树时选择了一个生物中的旁系同源基因,或者选择的是不同物种中的同源基因,即直系同源,那么构建的都属于基因树。
我们视生殖隔离的产生为物种分化的标志,所以基因分化时间和物种分化时间不一致。基因树和物种树主要存在的差异有:①两物种的两个基因分化发生在物种形成事件之前,导致对系统发育树中最长分支的分析过估计;②基因树的拓扑结构可能和物种树存在差异。
基于上述原因,只能通过基因树来推测物种树。研究人员构建物种树时往往利用多个基因或蛋白级联建树。
小tips:两个同源序列是直系还是旁系?
直系同源()不同物种中,来自物种分化。功能相似。
旁系同源()同一物种中,由于基因复制、累积变异产生。功能不同。
异源同源()不同物种中,基因的横向转移,比如溶菌酶基因GH25。功能相同。
硬要较个真儿的话,其实我们说不准,不同物种中的两个同源序列是直系还是旁系,比如A1和B2就是旁系同源。
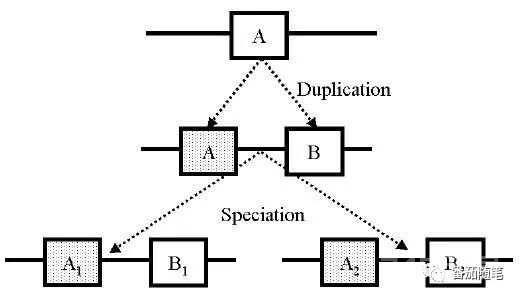
理论上,想要区分它们是很难的,只有通过一些判断,推测它们是哪一种。不能保证100%正确,但是起码我们把它们看作直系同源来建树也不会错得离谱。
推测方法:当我们有两个物种C和D的全基因组序列数据、蛋白质组大小相似的情况下,如果将C中序列C1与D中D1, D2, D3......Dn进行BLAST,返回结果的top1是D1;再反过来将D1与C1, C2, C3......Cn进行BLAST,返回结果top1是C1时,可以推测C1和D1应该是直系同源基因。
用类似且更复杂的推测方法建立了一个直系同源序列的集合——COG( of )。
(3)残基替换饱和度检验(Test of )
生物在进化发育中,nt/aa序列的残基会发生替换,通过对nt/aa残基替代数计算获得进化距离,而系统发育树是在进化距离基础上构建的。所以有必要对即将构建进化树的序列进行替换饱和度检验。
替换饱和度指的是一条序列同一位点残基发生多次替换(替换了几次又变成一样的残基了),或不同序列发生相同的替换。这会导致原本应有很大差异的两个序列,计算后却进化距离很近,从而导致长枝吸引现象(long ,LBA)。
如果序列替换趋向于饱和,那么包含的系统发育信息少,用这些序列建树的意义不大。很多时候我们直接建的树感觉没啥大问题,常忽略饱和度检验。
操作:残基替换饱和度检验
可以使用DAMBE软件进行分析(下载链接):
首先导入比对后的序列,进行seq.-> -> test by xia et al的分析。
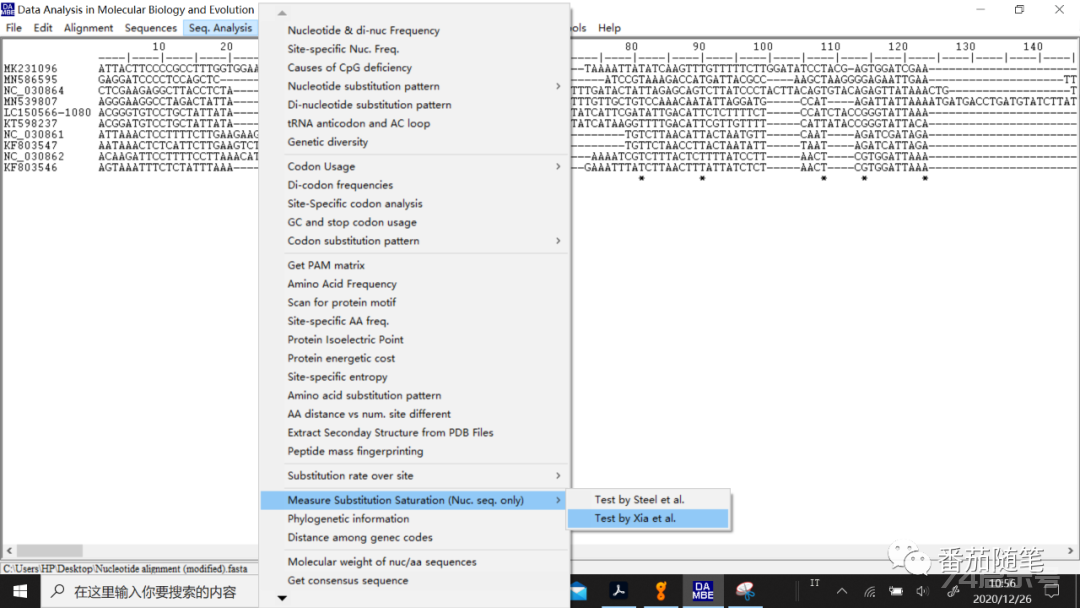
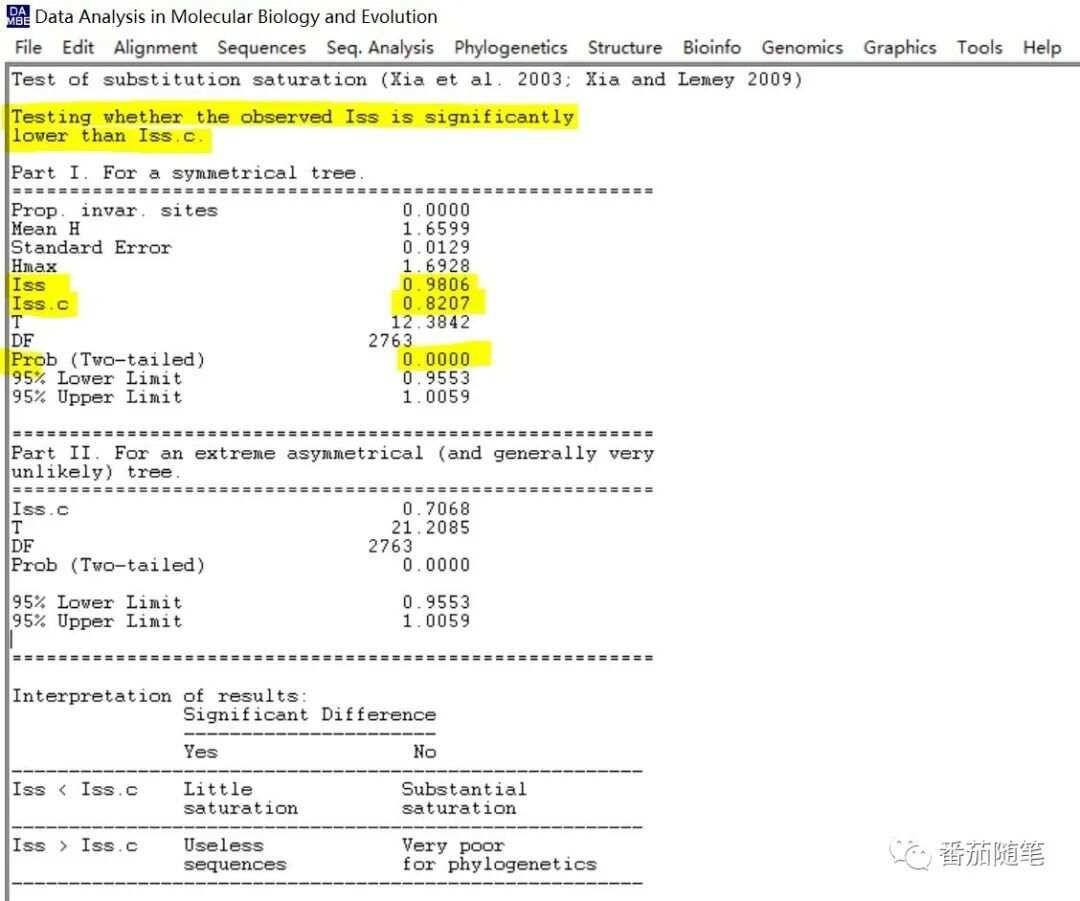
当Iss
(4)其他建树序列选择的tips
避免使用不完整的序列片段开始多序列比对;
避免使用异源同源序列;
避免使用重组序列。
小tips:常见的问题和可能原因
NCBI-BLAST中相似度最高的两个序列,建树的距离特别远?或者相似度很高的一组序列,系统发育树 (步长值)很低?
可能是:
(1)比对时,下载的序列A的第一个残基比对的序列B的最后一个残基,比对时序列方向需要手动调整。
(2)MSA质量太高,所有序列的相似度都很高,就很可能出现步长值低这样的情况。可以这样理解,因为序列间没有足够大的差异,所以没有计算出显著的建树结果。可以引入一些差异较大的序列、外群或换一种建树方法( → ;除了MEGA-X,还可以试试PhyML 3.0等网页)。
PhyML 3.0:
(3)MSA质量太低,检查所选的序列,或者从算法上可以考虑MP法进行建树。
2. 多序列比对结果编辑
多序列比对结果的质量是影响建树质量的关键因素。应使其尽量:
(1)没有大量gap
(2)剪辑MSA不齐的两端
(3)保守区域选择:即优化MSA质量,保证比对结果中含有信息量大的板块,使比对质量高,但是残基一致性低。适用于信息位点足够多的长序列。
根据以上原则(1)(2),图片中短序列MSA结果中三个框的部分就可以删除。
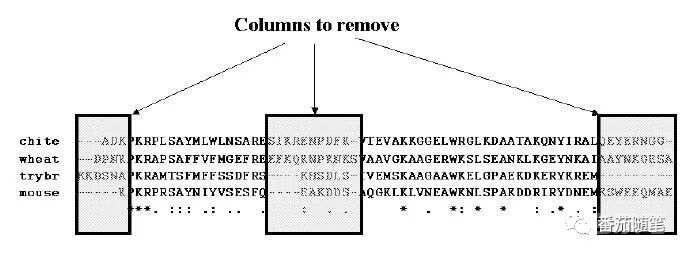
操作:使用选择保守区
线上工具:
还有本地版可以下载
有适合MAC,和Linux的不同版本。
一般这个操作都是针对长序列,但是为了节约时间,下面我用短的多序列比对结果举例。

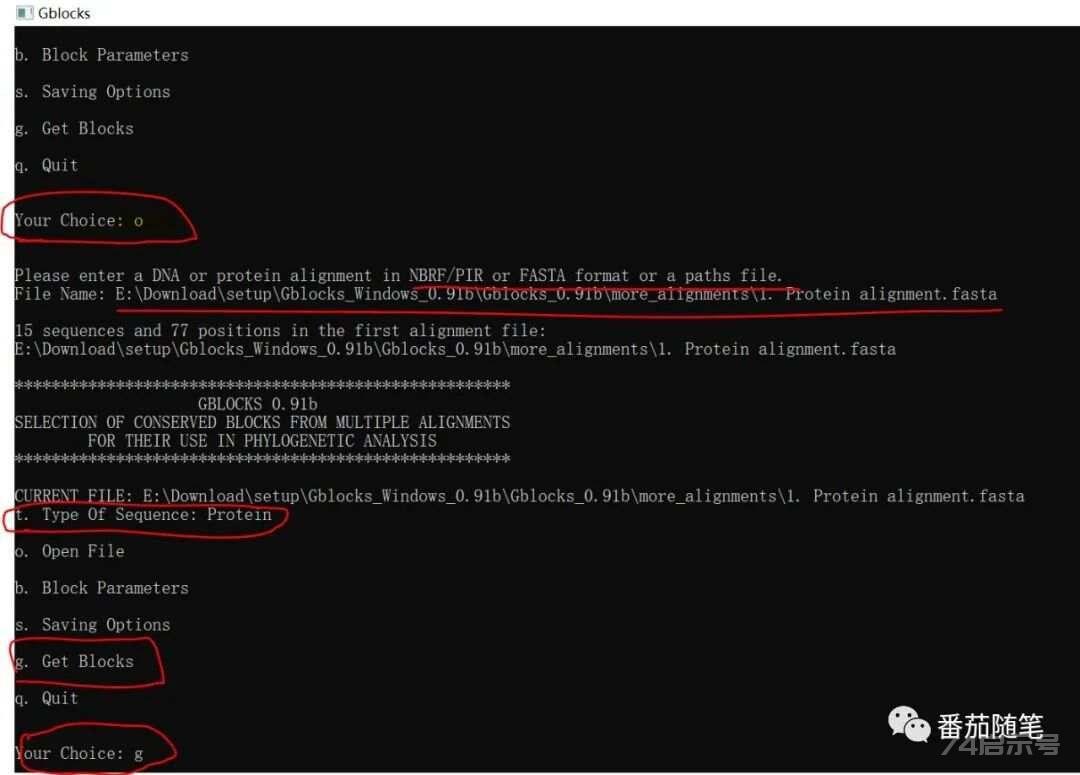
下载之后界面如下图,首先输入b然后回车,即设置Block →这里的5,即“ Gap ”有“None/Half/All”不同选择,默认是None,如果要更改就输入5再回车。根据分析要求选择不同的程度,我这里选了None→输入m回车,即打开主功能页面→输入o回车,即打开文件口令→然后输入文件地址加扩展名如下图→打开文件后,软件默认是蛋白质序列,如果不是可以输入t重选,我这里举例序列是蛋白质,就按照默认值,输入g回车获得保守区。
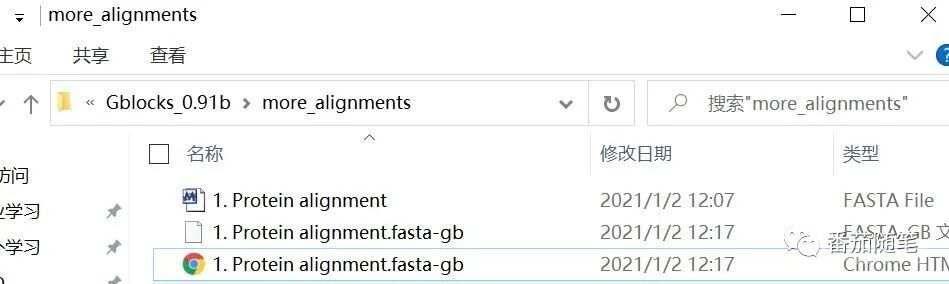
这时软件文件夹中会生成两个文件,一个是.fasta-gb文件(默认扩展名,可修改为fasta进行后续分析),另一个是网页文件,打开后标蓝色区域为保守区。

三、构建系统发育树
1. 不同建树方法
建树原理主要分为基于距离的方法,即输入距离矩阵即可的距离法;以及基于序列信息的方法,即简约法、似然法等。常见的方法简单介绍如下(更详细的原理有空再写,挖个坑):
(1)邻接法(-,NJ):基于最小进化原理,通过比较每对序列的距离,构建一个总距离最小的拓扑树。速度最快,结果较为准确,比较常用;更适用于进化距离不大(0
上一篇
“土法炼钢”的精神基因



