TCGA临床数据(化疗数据、用药反应等)和生存信息(4种临床结局)
- 科技
- 5个月前
- 31
专注R语言在生物医学中的使用
设为“星标”,精彩不错过
GDC官网中有3种来源的TCGA临床信息:
-和XML中的临床信息主要有两个区别:
下载的对象中的临床数据是 data,但是和直接使用得到的临床信息(这个也是 data)不太一样,并且还会添加一些从相关文献中得到的信息(这部分信息会添加前缀)。
从SE对象中提取的临床信息
我们以TCGA-COAD为例进行演示。
下面是从TCGA-COAD原始的对象(通过::("TCGA-COAD")下载的)提取的临床信息:
load("G:/easyTCGA_test/output_mRNA_lncRNA_expr/TCGA-COAD_clinical.rdata")
dim(clin_info)
##[1]524109
#colnames(clin_info)109列
一共109列临床信息,还是挺多的,但是很多信息平常都用不到。而且并不是所有的癌症都是109列,因为每个癌症的信息是不一样的,比如乳腺癌就会有分型信息(PAM50分型)。
这里面并没有用药信息、化疗信息、用药反应等。
下面我们通过下载临床数据。
library(TCGAbiolinks)
data
通过以下方式可以下载 data:
clinical.indexed<-GDCquery_clinic(project="TCGA-COAD",type="clinical")
dim(clinical.indexed)
##[1]46170
#colnames(clinical.indexed)
一共70列,虽然列名中有一些和化疗药物、放疗有关,但是并没有用药信息(可以自己点开看看)。
这个 data是从XML整理出来的,虽然没有XML全,但是好在信息都是最新的,而且里面的病理分期是分开的(TNM)是3列数据,XML中的病理分期是在一起的。所以通常来说这个临床信息是最好用的(除了没有药物等信息基本没啥缺点).
所以这个数据其实可以和XML中的数据结合使用,提取里面好用的即可。
这种方法得到的临床信息和从对象中提取的临床信息也不是完全一样(如下所示),但是比较关键的信息都是有的,比如病理分期、生存状态、生存时间等:
#取个交集看看
intersect(colnames(clin_info),colnames(clinical.indexed))
[1]"submitter_id""state"
[3]"synchronous_malignancy""ajcc_pathologic_stage"
[5]"days_to_diagnosis""last_known_disease_status"
[7]"tissue_or_organ_of_origin""days_to_last_follow_up"
[9]"age_at_diagnosis""primary_diagnosis"
[11]"prior_malignancy""year_of_diagnosis"
[13]"prior_treatment""ajcc_staging_system_edition"
[15]"ajcc_pathologic_t""morphology"
[17]"ajcc_pathologic_n""ajcc_pathologic_m"
[19]"classification_of_tumor""diagnosis_id"
[21]"icd_10_code""site_of_resection_or_biopsy"
[23]"tumor_grade""progression_or_recurrence"
[25]"alcohol_history""exposure_id"
[27]"race""gender"
[29]"ethnicity""vital_status"
[31]"age_at_index""days_to_birth"
[33]"year_of_birth""demographic_id"
[35]"days_to_death""bcr_patient_barcode"
[37]"year_of_death"
XML临床信息
可以通过官网下载XML格式的临床信息,点点点即可,如下图所示,官网的选择结果:
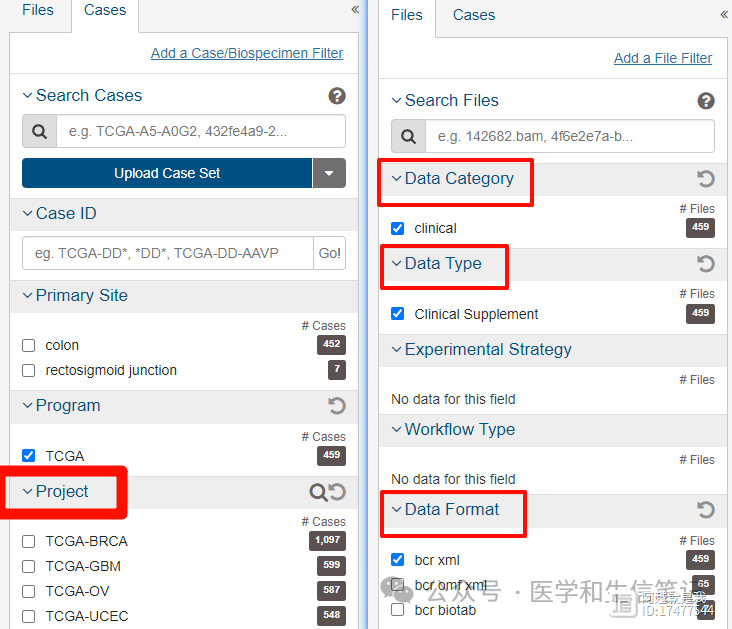
当然也可以通过下载XML格式的临床数据,但是要注意,由于一个病人可能有多个临床信息(比如有多次化疗信息),所以一次只能解析一个表格,需要通过参数指定要解析的数据:
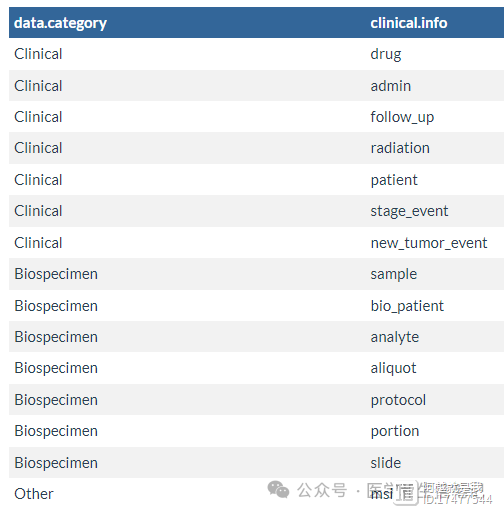
使用下载官网的XML临床信息,注意这种方式其中的几个参数和官网都是一一对应的,所以数量啥的都是和官网完全一致的:
#下载XML临床数据
query1<-GDCquery(
project="TCGA-COAD",
data.category="Clinical",
data.type="ClinicalSupplement",
data.format="bcrxml"
)
##--------------------------------------
##oGDCquery:SearchinginGDCdatabase
##--------------------------------------
##Genomeofreference:hg38
##--------------------------------------------
##ooAccessingGDC.Thismighttakeawhile...
##--------------------------------------------
##oooProject:TCGA-COAD
##--------------------
##ooFilteringresults
##--------------------
##oooBydata.format
##oooBydata.type
##----------------
##ooCheckingdata
##----------------
##oooCheckingifthereareduplicatedcases
##oooCheckingifthereareresultsforthequery
##-------------------
##oPreparingoutput
##-------------------
GDCdownload(query1)
##DownloadingdataforprojectTCGA-COAD
##GDCdownloadwilldownload459files.Atotalof17.981211MB
##Downloadingas:Wed_Jan_10_20_02_01_2024.tar.gz
#解析patient信息,指定clinical.info
clinical.patient.xml<-GDCprepare_clinic(query1,clinical.info="patient")
##Togetthefollowinginformationpleasechangetheclinical.infoargument
##=>new_tumor_events:new_tumor_event
##=>drugs:drug
##=>follow_ups:follow_up
##=>radiations:radiation
##Parsingfollowupversion:follow_up_v1.0
##Addingstageeventinformation
##Updatingdays_to_last_followupandvital_statusfromfollow_upinformationusinglastentry
##Parsingfollowupversion:follow_up_v1.0
dim(clinical.patient.xml)#也是459,和上面的图片一样
##[1]45976
#colnames(clinical.patient.xml)
这里面的很多信息和上面两种是重复的,毕竟上面两种都是从XML中提取的。而且这个信息看似很全,但是很多信息都是NA,比如其中的生存信息就是不全的,大部分都是NA.
解析药物信息,这里面就有各种化疗药物及用药反应信息:
clinical.drug.xml<-GDCprepare_clinic(query1,clinical.info="drug")
colnames(clinical.drug.xml)
##[1]"bcr_patient_barcode""tx_on_clinical_trial"
##[3]"regimen_number""bcr_drug_barcode"
##[5]"bcr_drug_uuid""total_dose"
##[7]"total_dose_units""prescribed_dose"
##[9]"prescribed_dose_units""number_cycles"
##[11]"days_to_drug_therapy_start""days_to_drug_therapy_end"
##[13]"therapy_types""drug_name"
##[15]"clinical_trail_drug_classification""regimen_indication"
##[17]"regimen_indication_notes""route_of_administrations"
##[19]"therapy_ongoing""measure_of_response"
##[21]"day_of_form_completion""month_of_form_completion"
##[23]"year_of_form_completion""project"
dim(clinical.drug.xml)
##[1]59324
#用药信息和药物反应,CP,PR等
clinical.drug.xml[156:160,c("drug_name","measure_of_response")]
##drug_namemeasure_of_response
##156LeucovorinCompleteResponse
##157FluorouracilCompleteResponse
##158FluorouracilCompleteResponse
##159FolinicacidPartialResponse
##1605-FluorouracilPartialResponse
解析信息,这个信息和无病生存期有关(但是这个我们有更好的选择,用TCGA-CDR的数据):
clinical.nte.xml<-GDCprepare_clinic(query1,clinical.info="new_tumor_event")
#colnames(clinical.nte.xml)
dim(clinical.nte.xml)
##[1]11810
解析随访信息,这里面的随访数据是最新的数据,但其实也是很久没更新过了,-中的随访信息就是从这里更新.但是这里面有很多重复的信息,比如某个人有多次随访,都会记录在这里,如果你自己整理的话就很浪费时间.
clinical.followup.xml<-GDCprepare_clinic(query1,clinical.info="follow_up")
##Parsingfollowupversion:follow_up_v1.0
#colnames(clinical.followup.xml)
dim(clinical.followup.xml)
##[1]57718
这里面也有、药物反应(CR/PR等)等信息。
如果需要最原始的XML信息,可以从XML文件中提取,如果要整理好的数据,可以使用-,这样看这个就显得不是那么重要了。
下载BCR 临床信息可以使用以下代码:
query<-GDCquery(
project="TCGA-COAD",
data.category="Clinical",
data.type="ClinicalSupplement",
data.format="bcrbiotab"
)
GDCdownload(query)
clinical.BCRtab.all<-GDCprepare(query)#结果是7,也是和上面图片一样的
##Newnames:
##·`history_other_malignancy`->`history_other_malignancy...11`
##·`history_other_malignancy`->`history_other_malignancy...44`
#看看包含哪些信息
names(clinical.BCRtab.all)
##[1]"clinical_nte_coad""clinical_patient_coad"
##[3]"clinical_omf_v4.0_coad""clinical_follow_up_v1.0_nte_coad"
##[5]"clinical_drug_coad""clinical_radiation_coad"
##[7]"clinical_follow_up_v1.0_coad"
有随访信息,也有放化疗信息等,但是和XML中的也不是完全一样哈。
简单看下信息:
colnames(clinical.BCRtab.all[["clinical_nte_coad"]])
##[1]"bcr_patient_uuid"
##[2]"bcr_patient_barcode"
##[3]"new_tumor_event_dx_days_to"
##[4]"new_tumor_event_site_surgery"
##[5]"new_tumor_event_radiation_tx"
##[6]"new_tumor_event_pharmaceutical_tx"
##[7]"days_to_new_tumor_event_additional_surgery_procedure"
##[8]"new_neoplasm_event_occurrence_anatomic_site"
##[9]"new_neoplasm_event_type"
##[10]"new_neoplasm_occurrence_anatomic_site_text"
##[11]"new_tumor_event_additional_surgery_procedure"
##[12]"progression_determined_by"
##[13]"residual_disease_post_new_tumor_event_margin_status"
通过以上简单的探索发现,XML中的信息无疑是最全的,但是也是最原始的,原始意味着对初学不友好,需要很多自己整理的过程,此时 data是更好的选择,因为是经过整理过的。
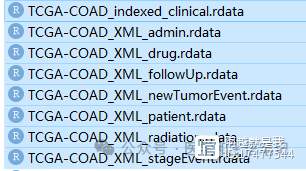
我在包中添加了一个函数,可以1行代码下载XML以及 data,并且会自动帮你提取其中的数据,并保存为rdata格式。
比如使用如下1行代码:
getclinical("TCGA-COAD")
即可得到以下数据:
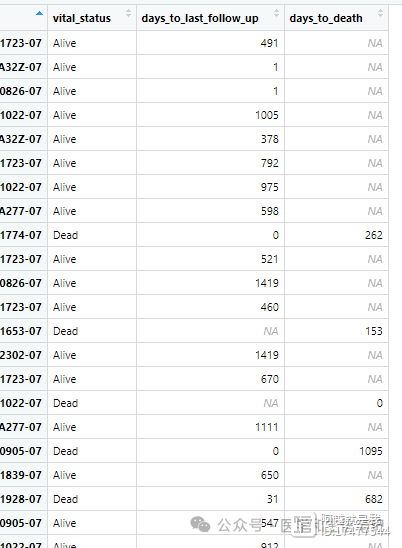
生存时间用哪个
生存时间用哪个?up还是?
可以看到当是Alive时,up是有时间的,此时是NA,当是Dead时,up是NA,此时是有时间的。
目前很多人都在用的做法是二者相加。但是也有人发现有些会同时有两列时间,比如:
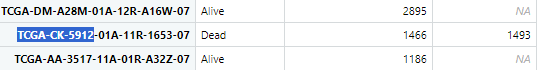
这个结果是从SE对象中提取出来的,我又看了下XML( = "")中的信息,发现此时只有是有时间的:

所以我做了如下处理,如果生存状态是Dead,就使用的时间,如果是Alive,就使用up的时间。
我把这个结果和从XENA下载的结果比较了一下,发现是正确的:

整理一下SE对象中的生存信息:
library(tidyverse)
coad_surv_gdc<-clin_info%>%
select(sample_submitter_id,patient,vital_status,days_to_last_follow_up,
days_to_death)%>%
mutate(OS_time=if_else(vital_status=="Dead",days_to_death,
days_to_last_follow_up),
vital_status=if_else(vital_status=="Dead",1,0))%>%
select(sample=sample_submitter_id,OS=vital_status,
`_PATIENT`=patient,OS.time=OS_time
)
rownames(coad_surv_gdc)<-NULL
coad_surv_gdc%>%distinct(`_PATIENT`,.keep_all=T)%>%dim()
##[1]4584
head(coad_surv_gdc)
##sampleOS_PATIENTOS.time
##1TCGA-D5-6540-01A0TCGA-D5-6540491
##2TCGA-AA-3525-11A0TCGA-AA-35251
##3TCGA-AA-3525-01A0TCGA-AA-35251
##4TCGA-AA-3815-01A0TCGA-AA-38151005
##5TCGA-D5-6923-01A0TCGA-D5-6923378
##6TCGA-G4-6322-01A0TCGA-G4-6322792
这个可能也不是完全正确,但是我也找不到官方的说法到底用哪个,毕竟用啥的都有,我之前一直都是用的up......如果大家知道更好的方式,欢迎告诉我。
把从XENA下载的生存数据读取进来:
coad_surv_xena<-data.table::fread("../000files/TCGA-COAD.survival.tsv",
data.table=F)
names(coad_surv_xena)
##[1]"sample""OS""_PATIENT""OS.time"
dim(coad_surv_xena)
##[1]5394
coad_surv_xena%>%distinct(`_PATIENT`,.keep_all=T)%>%dim()
##[1]4374
head(coad_surv_xena)
##sampleOS_PATIENTOS.time
##1TCGA-AA-3492-01A1TCGA-AA-34921
##2TCGA-AA-3492-11A1TCGA-AA-34921
##3TCGA-G4-6626-01A1TCGA-G4-66261
##4TCGA-AA-3525-01A0TCGA-AA-35251
##5TCGA-AA-3525-11A0TCGA-AA-35251
##6TCGA-AY-6196-01A0TCGA-AY-61966
取个交集看看:
#新发现:inner_join不会自动去重,intersect会自动去重!
common_surv<-inner_join(coad_surv_gdc%>%
distinct(`_PATIENT`,.keep_all=T),
coad_surv_xena%>%
distinct(`_PATIENT`,.keep_all=T),
by=c("sample","OS","OS.time"))
dim(common_surv)
##[1]3815
381个相同的,效果还是不错的。
探索下不同的那些。
diff_surv1<-setdiff(coad_surv_gdc%>%
distinct(`_PATIENT`,.keep_all=T),
coad_surv_xena%>%
distinct(`_PATIENT`,.keep_all=T))%>%
drop_na()%>%
filter(OS.time>0)#在gdc,不在xena
diff_surv2<-setdiff(coad_surv_xena%>%distinct(`_PATIENT`,.keep_all=T),
coad_surv_gdc%>%distinct(`_PATIENT`,.keep_all=T))#在xena,不在gdc
TCGA-PAN CDR
GDC官方发表在cell上的文章:An TCGA Pan- Data to Drive High-
这篇文章提取出了4种TCGA的随访结局(详情自己读文章):
这个结果可以直接下载:
也可以通过XENA()下载,但是两者略有区别(不影响使用)。
我们下载XENA的,读取进来看看:
tcga_pan_surv<-data.table::fread("G:/bioinfo/000files/Survival_SupplementalTable_S1_20171025_xena_sp",data.table=F)
#筛选TCGA-COAD的
tcga_pan_surv%>%
filter(`cancertypeabbreviation`=="COAD")%>%
select(1,2,26:33)
##sample_PATIENTOSOS.timeDSSDSS.timeDFIDFI.timePFI
##1TCGA-3L-AA1B-01TCGA-3L-AA1B0475047504750
##2TCGA-4N-A93T-01TCGA-4N-A93T01460146NANA0
##3TCGA-4T-AA8H-01TCGA-4T-AA8H0385038503850
##4TCGA-5M-AAT4-01TCGA-5M-AAT4149149NANA1
##5TCGA-5M-AAT6-01TCGA-5M-AAT612901290NANA1
##6TCGA-5M-AATE-01TCGA-5M-AATE0120001200NANA1
##7TCGA-A6-2670-01TCGA-A6-267007750775NANA0
##8TCGA-A6-2671-01TCGA-A6-26711133111331NANA1
##9TCGA-A6-2671-11TCGA-A6-26711133111331NANA1
##PFI.time
##1475
##2146
##3385
##449
##5219
##6810
##7775
##8535
##9535
结果直接给出了各种随访结局和时间,其中OS和OS.time这两个信息和GDC的差别不大(充分说明GDC官方每次更新的都不是这些内容)。
所以我把这个结果直接整合进中了,方便大家使用,这样如果是OS,那么可以用GDC整理的,也可以用这个,如果是DSS/DFI/PFI,就直接用这个就好了。
可通过以下方式加载:
library(easyTCGA)#安装最新版本
data("tcga_cdr_surv")
dim(tcga_cdr_surv)
##[1]1259134
tcga_cdr_surv[1:6,26:33]
##OSOS.timeDSSDSS.timeDFIDFI.timePFIPFI.time
##1113551135517541754
##21167711677NANA1289
##30209102091153153
##414231423NANA1126
##513651365NANA150
##602703027030270302703
参考资料官方文档:GDC官方文档:#-data