胃癌单细胞数据集GSE163558复现(十):CopyKAT分析
- 健康养生
- 9小时前
- 40
前言
Hello小伙伴们大家好,我是生信技能树的小学徒”我才不吃蛋黄“。今天是胃癌单细胞数据集复现系列第十期。第九期我们用TCGA-STAD数据进行生存分析。本期,我们将回到高级分析(分析),通过计算CNV评估上皮细胞良恶性。
1.背景介绍
在第六期推文中,我们联合TCGA-STAD数据给上皮添加了恶性评分,从而协助区分肿瘤细胞与非肿瘤细胞。在高级分析中,我们可以利用和对单细胞进行CNV(Copy , 拷贝数变异)分析,区分肿瘤细胞与非肿瘤细胞。
CNV是基因组结构变异( ,SV)的重要组成部分,主要原因是基因组发生重排,一般指长度为1kb以上的基因组大片段的拷贝数增加或者减少,主要表现为亚显微水平的缺失和重复。CNV在肿瘤的发生和发展研究中扮演重要的角色。
本期我们使用 ( of )进行CNV分析,它是一种集成贝叶斯分割方法。利用scRNA-seq数据推断人类肿瘤的基因组拷贝数,然后通过对拷贝数数据进行分层聚类,以识别非整倍体肿瘤细胞和二倍体基质细胞之间的最大距离。其原理同样是通过单细胞转录组数据来推断细胞的染色体倍数,进而推断是正常细胞()还是肿瘤细胞()。
2.数据分析2.1 导入数据
首先清除系统环境变量,加载R包,设置新文件夹和工作目录,导入恶性上皮细胞数据:
rm(list=ls())
options(stringsAsFactors=F)
library(Seurat)
library(ggplot2)
library(clustree)
library(cowplot)
library(dplyr)
library(infercnv)
library(copykat)
library(tidyverse)
#devtools::install_github("broadinstitute/infercnv")
dir.create("8-CNV")
getwd()
setwd("../8-CNV")
sce=readRDS("../6-TCGA_STAD/malignant.rds")
table(sce$celltype)
Idents(sce)=sce$celltype
避免后面流程运行的太长了对细胞进行抽样:
seurat_object=sce
seurat_object<-subset(seurat_object,downsample=200)
table(Idents(seurat_object))
预测肿瘤/正常细胞状态的基本原理是非整倍体在人类癌症中很常见 (90%)。具有广泛全基因组拷贝数畸变(非整倍体)的细胞被认为是肿瘤细胞,而基质正常细胞和免疫细胞通常具有2N二倍体或接近二倍体的拷贝数分布。通过单细胞转录组数据来推断细胞的染色体倍数,进而推断是正常细胞()还是肿瘤细胞()。它还可以进一步对肿瘤细胞进行聚类,找出不同的亚群。
运行
ngene.chr参数是过滤细胞的一个标准,它要求被用来做CNV预测的细胞,一个染色体上至少有5个基因。
sam.name定义样本名称 ( name),会给出来的文件加前缀
scRNA<-seurat_object
counts<-as.matrix(scRNA@assays$RNA$counts)
table(scRNA$celltype)
if(T){cnv<-copykat(rawmat=counts,ngene.chr=5,sam.name="test",n.cores=8)
saveRDS(cnv,"cnv.rds")}
添加结果到对象meta.data信息中:
cnv=readRDS("cnv.rds")
table(rownames(cnv$CNAmat))
a=cnv$prediction$copykat.pred
table(a)
scRNA$CopyKAT=a
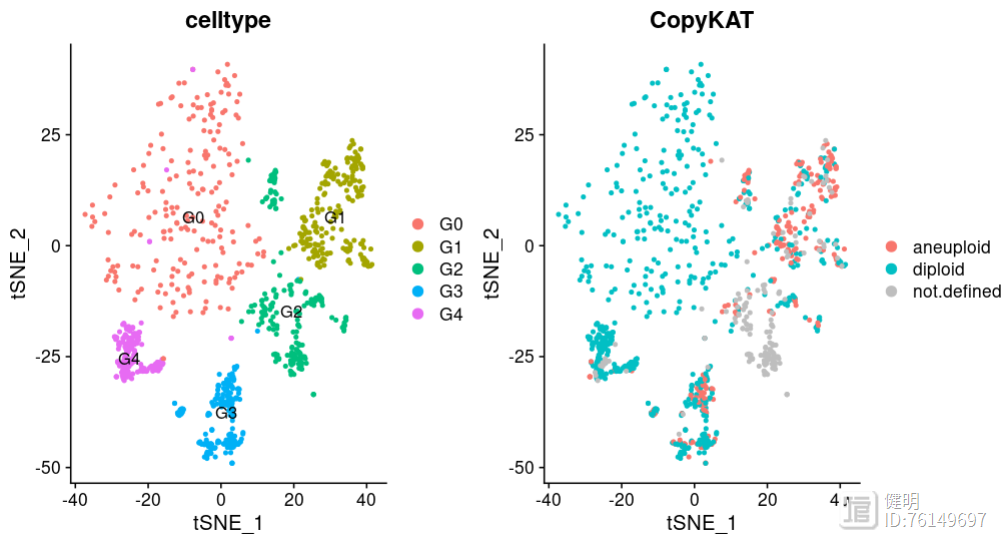
结果可视化:正常细胞(,蓝色)还是肿瘤细胞(,红色)
p1<-DimPlot(scRNA,group.by="celltype",label=T,reduction='tsne')
p2<-DimPlot(scRNA,group.by="CopyKAT",reduction='tsne')+scale_color_manual(values=c("#F8766D",'#02BFC4',"gray"))
pc<-p1+p2
pc
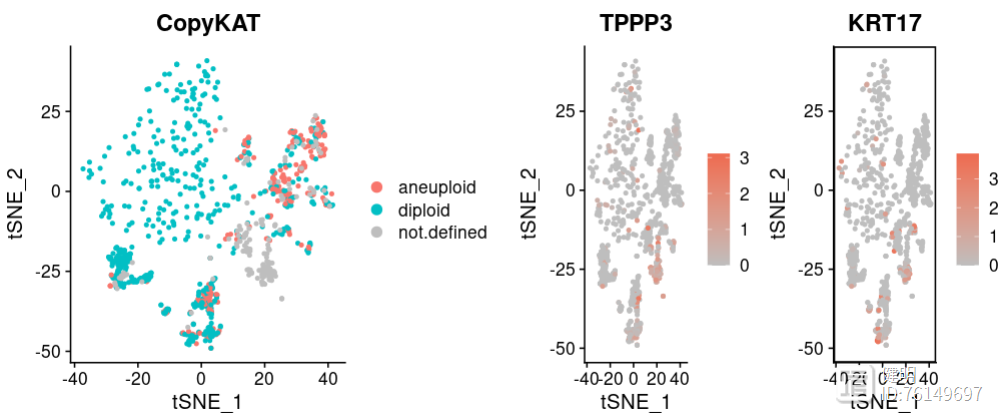
可视化maker基因'TPPP3','KRT17'的表达情况,检查结果准确性:
cols=c("gray","coral2")
plot<-FeaturePlot(scRNA,features=c('TPPP3','KRT17'),cols=cols,pt.size=1,reduction='tsne')+
theme(panel.border=element_rect(fill=NA,color="black",size=1,linetype="solid"))#加边框
plot_grid(p2,plot)
结果主要有两个:
1)预测的结果正常细胞()还是肿瘤细胞();
2) 每个CNV 在每个细胞的表达量。这里看出和的不同来了,是基于 而不是gene level的表达量。
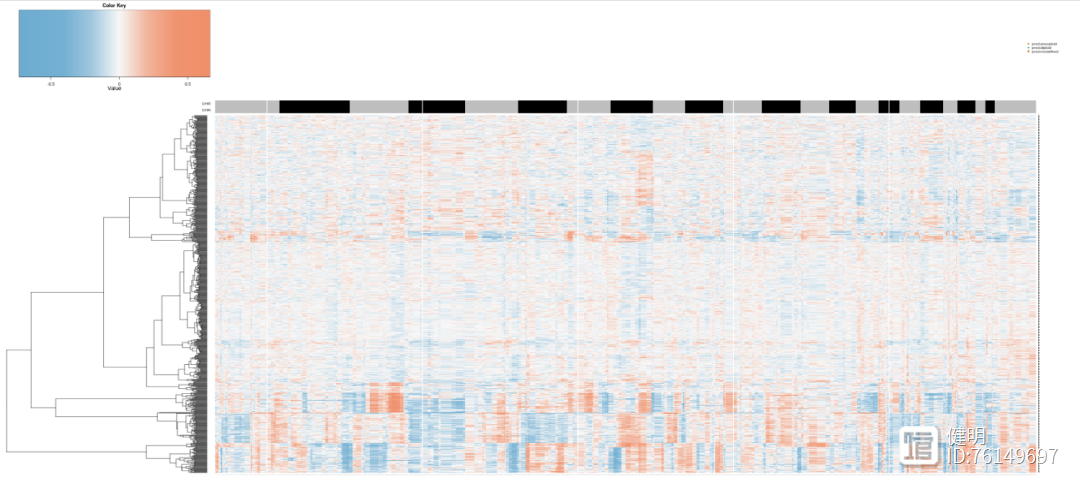
绘制热图
copykat.test=cnv
pred.test<-data.frame(copykat.test$prediction)
CNA.test<-data.frame(copykat.test$CNAmat)
head(pred.test)
head(CNA.test[,1:5])
my_palette<-colorRampPalette(rev(RColorBrewer::brewer.pal(n=3,name="RdBu")))(n=999)
chr<-as.numeric(CNA.test$chrom)%%2+1
rbPal1<-colorRampPalette(c('black','grey'))
CHR<-rbPal1(2)[as.numeric(chr)]
chr1<-cbind(CHR,CHR)
rbPal5<-colorRampPalette(RColorBrewer::brewer.pal(n=8,name="Dark2")[2:1])
com.preN<-pred.test$copykat.pred
pred<-rbPal5(2)[as.numeric(factor(com.preN))]
cells<-rbind(pred,pred)
col_breaks=c(seq(-1,-0.4,length=50),seq(-0.4,-0.2,length=150),seq(-0.2,0.2,length=600),seq(0.2,0.4,length=150),seq(0.4,1,length=50))
heatmap.3(t(CNA.test[,4:ncol(CNA.test)]),dendrogram="r",distfun=function(x)parallelDist::parDist(x,threads=4,method="euclidean"),
hclustfun=function(x)hclust(x,method="ward.D2"),
ColSideColors=chr1,Colv=NA,Rowv=TRUE,
notecol="black",col=my_palette,breaks=col_breaks,key=TRUE,
keysize=1,density.info="none",trace="none",
cexRow=0.1,cexCol=0.1,cex.main=1,cex.lab=0.1,
symm=F,symkey=F,symbreaks=T,cex=1,cex.main=4,margins=c(10,10))
legend("topright",paste("pred.",names(table(com.preN)),sep=""),pch=15,col=RColorBrewer::brewer.pal(n=8,name="Dark2")[2:1],cex=0.6,bty="n")
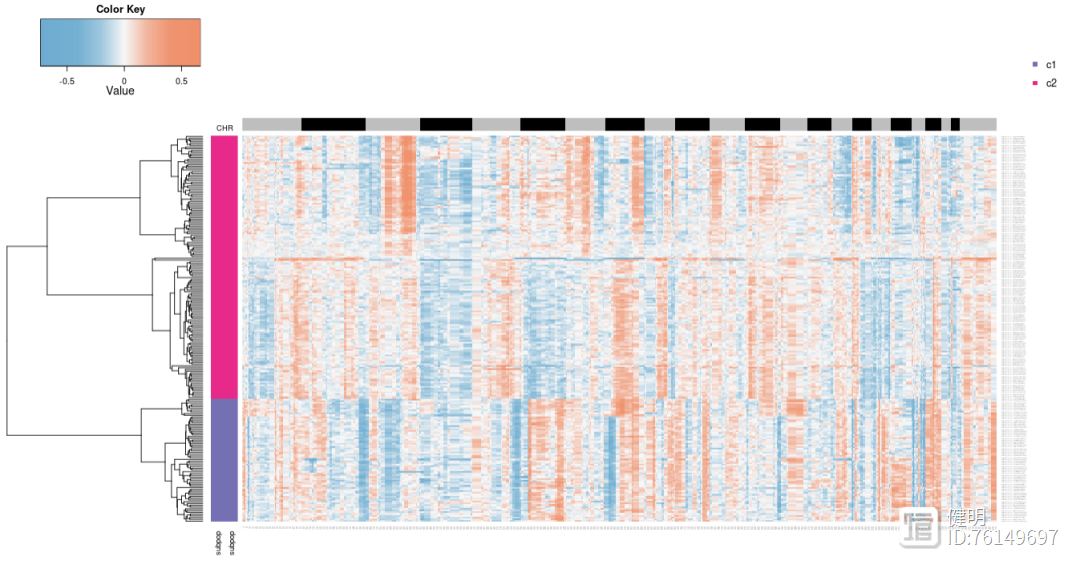
再对肿瘤细胞再聚类并画热图,又能分成两群。
table(pred.test$copykat.pred)
tumor.cells<-pred.test$cell.names[which(pred.test$copykat.pred=="aneuploid")]
colnames(CNA.test)<-gsub(".1$","-1",colnames(CNA.test))
tumor.mat<-CNA.test[,colnames(CNA.test)%in%tumor.cells]
hcc<-hclust(parallelDist::parDist(t(tumor.mat),threads=4,method="euclidean"),method="ward.D2")
hc.umap<-cutree(hcc,2)
rbPal6<-colorRampPalette(RColorBrewer::brewer.pal(n=8,name="Dark2")[3:4])
subpop<-rbPal6(2)[as.numeric(factor(hc.umap))]
cells<-rbind(subpop,subpop)
heatmap.3(t(tumor.mat),dendrogram="r",distfun=function(x)parallelDist::parDist(x,threads=4,method="euclidean"),
hclustfun=function(x)hclust(x,method="ward.D2"),
ColSideColors=chr1,RowSideColors=cells,Colv=NA,Rowv=TRUE,
notecol="black",col=my_palette,breaks=col_breaks,key=TRUE,
keysize=1,density.info="none",trace="none",
cexRow=0.1,cexCol=0.1,cex.main=1,cex.lab=0.1,
symm=F,symkey=F,symbreaks=T,cex=1,cex.main=4,margins=c(10,10))
legend("topright",c("c1","c2"),pch=15,col=RColorBrewer::brewer.pal(n=8,name="Dark2")[3:4],cex=0.9,bty='n')
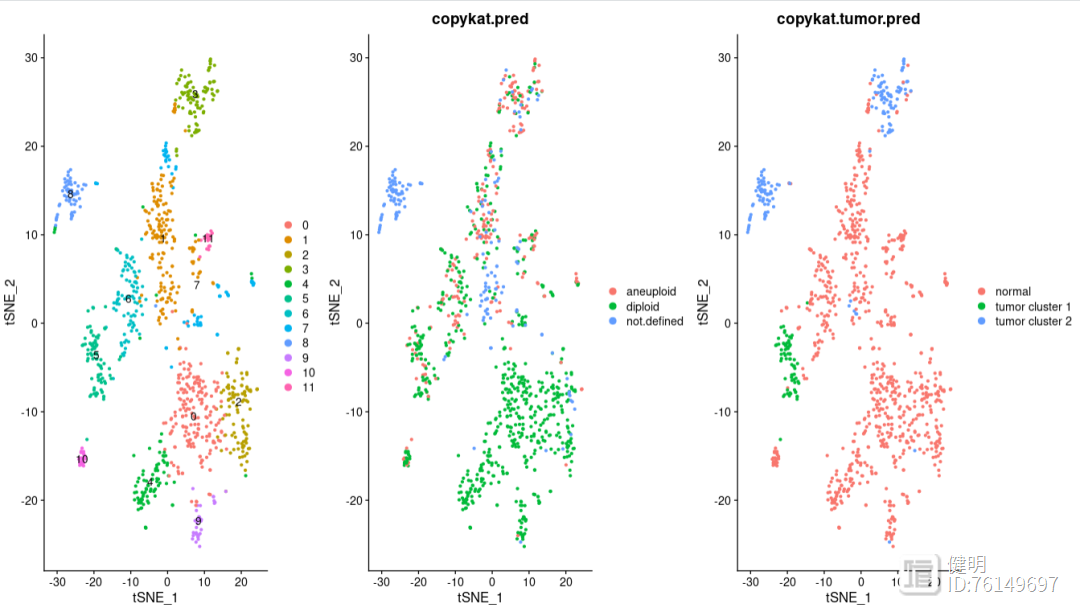
最后把CNV的结果投射到单细胞聚类结果上看一看是否合理,标准流程走一遍,聚类结果和分群结果投射到TSNE上。
standard10X=function(dat,nPCs=50,res=1.0,verbose=FALSE){
srat=CreateSeuratObject(dat)
srat=NormalizeData(srat,verbose=verbose)
srat=ScaleData(srat,verbose=verbose)
srat=FindVariableFeatures(srat,verbose=verbose)
srat=RunPCA(srat,verbose=verbose)
srat=RunTSNE(srat,dims=seq(nPCs),verbose=verbose)
srat=FindNeighbors(srat,dims=seq(nPCs),verbose=verbose)
srat=FindClusters(srat,res=res,verbose=verbose)
return(srat)
}
GC1<-standard10X(counts,nPCs=30,res=0.8)
GC1$copykat.pred<-pred.test$copykat.pred
GC1$copykat.tumor.pred<-rep("normal",nrow(GC1@meta.data))
table(hc.umap)
GC1$copykat.tumor.pred[rownames(GC1@meta.data)%in%names(hc.umap[hc.umap==1])]<-"tumorcluster1"
GC1$copykat.tumor.pred[rownames(GC1@meta.data)%in%names(hc.umap[hc.umap==2])]<-"tumorcluster2"
p1<-DimPlot(GC1,label=T)
p2<-DimPlot(GC1,group.by="copykat.pred")
p3<-DimPlot(GC1,group.by="copykat.tumor.pred")
p1+p2+p3
可以看到:2,4,8群是肿瘤细胞亚克隆
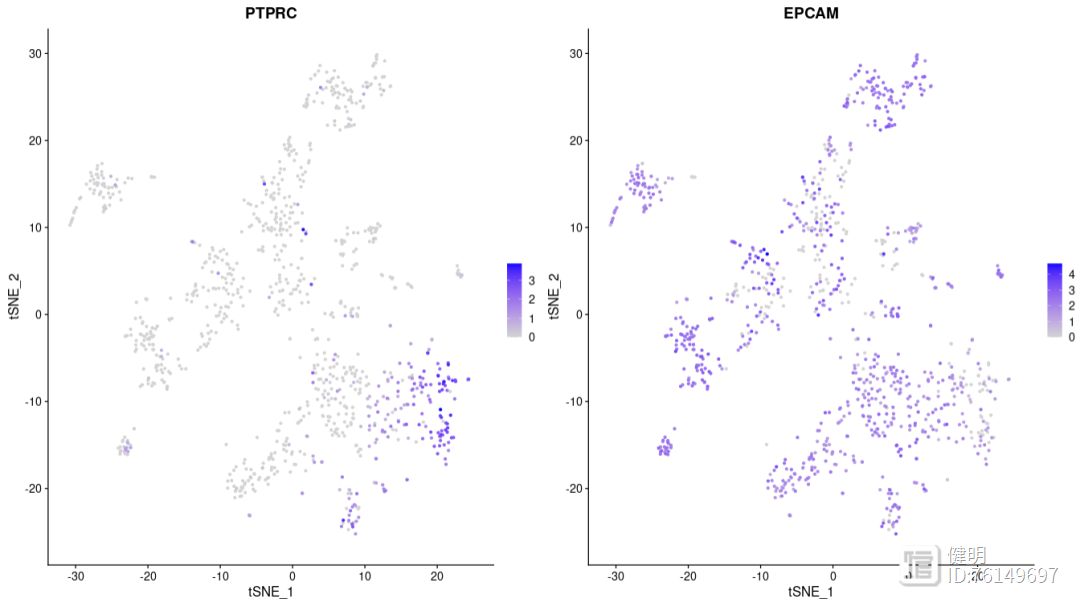
从免疫细胞和肿瘤细胞的标记基因表达来看,可以正确找出正常细胞和肿瘤细胞。
FeaturePlot(GC1,features=c("PTPRC","EPCAM"),order=T)
结语
本期,我们使用分析评估上皮细胞良恶性。下一期,我们将对T细胞亚群进行细分。顺便提前预告一下,胃癌系列推文完成后,将开启肺腺癌单细胞数据集复现系列,相关视频已经在B站上线:
文献推文详见(单细胞测序+空间转录组描绘从癌前病变到浸润性肺腺癌的动态演变 )。此外,关于推文内容的提升和优化,欢迎大家提宝贵意见。谢谢!







